
每3秒,全球就會多1位癡呆患者,其中約60%~80%會被確診為阿爾茨海默病(AD)。而在中國,AD患者已經(jīng)超過1000萬人,醫(yī)療費用高達每年1.6萬億元。
提高AD早期就診率,抓住黃金干預時間窗,是業(yè)內(nèi)公認的AD診療策略。今年7月,復旦大學附屬華山醫(yī)院神經(jīng)內(nèi)科郁金泰教授團隊領銜,聯(lián)合復旦大學類腦智能科學與技術(shù)研究院的馮建峰/程煒團隊,在Nature Human Behavior發(fā)表論文揭開了一種新生物標志物YWHAG的面紗。
9月6日上午,在由每日經(jīng)濟新聞主辦的2024 Inclusion·外灘大會見解論壇“AI醫(yī)藥革命:產(chǎn)業(yè)鏈創(chuàng)新之路”上,復旦大學類腦智能科學與技術(shù)研究院研究員程煒對相關研究進行了詳盡介紹。他表示,新型標志物的挖掘和早期識別離不開大數(shù)據(jù)和AI算法的助力,“一管血測AD”的時代已經(jīng)近了。
 圖片來源:主辦方提供
圖片來源:主辦方提供
作為一種連續(xù)發(fā)展長達10年~20年的老年疾病,阿爾茨海默病的早期干預是治療關鍵。根據(jù)2023年發(fā)表在Nature Human Behaviour的一項研究,如果在AD早期借助藥物或訓練調(diào)節(jié)既往病史、生活方式、身體指標等可控因素,可降低47%~72.6%的AD的發(fā)生。
但在國內(nèi),AD患者的臨床早期就診率只有14%。程煒表示,這是因為AD起病隱匿,各階段生物學指標演化規(guī)律尚不清晰,早期風險識別模型的準確率與解釋性不足,導致AD的早期識別和預測非常困難,而這也在很大程度上影響了單抗藥物等AD治療藥物的療效。
因此,獲取AD患者的非臨床數(shù)據(jù),構(gòu)建早期風險識別模型,是AI輔助AD早期預防和診治的關鍵,其中大數(shù)據(jù)和算法是兩塊重要拼圖。
程煒介紹,目前,國內(nèi)外圍繞AD建立了大量的隊列數(shù)據(jù)庫。例如,英國的UK Biobank是一個涵蓋社區(qū)人群的大型數(shù)據(jù)庫,跟蹤了約50萬名35歲~50歲參與者近20年的生活方式和生化指標;美國的All of Us則囊括了約100萬名社區(qū)成員,通過結(jié)合體檢機構(gòu)和臨床樣本的數(shù)據(jù),構(gòu)建了一個龐大的美國人群健康隊列。
參照國外的研究,中國也在衰老和癡呆社區(qū)隊列上進行了大量投入。目前,由復旦大學附屬華山醫(yī)院神經(jīng)內(nèi)科郁金泰教授領導的科技創(chuàng)新2030——“腦科學與類腦研究”重大項目,已經(jīng)依托華山醫(yī)院在全國范圍內(nèi)建立了社區(qū)腦健康衰老隊列(head隊列),旨在在中國不同地區(qū)招募2萬名參與者,通過長期隨訪進行縱向研究,描繪個體從健康狀態(tài)逐漸發(fā)展到癡呆癥狀的全過程。
此外,AI算法在老年腦疾病智能診療、推動老年健康領域取得了顯著進展。例如,結(jié)合AI算法和醫(yī)院病例數(shù)據(jù),可以實現(xiàn)對患者復發(fā)的有效預測;通過多模態(tài)數(shù)據(jù)的融合,應用深度學習算法,可以對不同AD亞型做出精準預測,精度超過臨床專家。
“有了大數(shù)據(jù),也有了AI算法,我們做的就是通過它們發(fā)現(xiàn)AD診斷新型標志物。”程煒表示,AD從無癥狀期發(fā)展到有癥狀期大約需要20年,而腦脊液生物標志物在AD病程中最先發(fā)生變化,因此團隊的首個研究工作是聚焦大規(guī)模人群的腦脊液蛋白質(zhì)組數(shù)據(jù),并基于這些數(shù)據(jù)開發(fā)出一種生成分析算法,識別與AD相關的蛋白質(zhì)。
在這一過程中,團隊從數(shù)千種腦脊液蛋白質(zhì)中篩選出多個與AD顯著相關的生物標志物,發(fā)現(xiàn)YWHAG、SMOC1、PIGR與TMOD2是AD診斷最重要的蛋白。根據(jù)四個蛋白構(gòu)建的AI模型,四個蛋白在AUC(此值越接近1,模型的診斷能力越好)上達到0.987的高準確度,超過了傳統(tǒng)的Aβ42生物標志物;即便只使用YWHAG單個蛋白,也能實現(xiàn)0.97的準確率。
為了驗證該模型的泛化能力,研究者還將其應用到尸檢病理隊列和獨立外部隊列中,發(fā)現(xiàn)上述蛋白仍能以高AUC值準確判斷個體是否患有AD,其中YWHAG在尸檢病理隊列和獨立外部隊列中診斷AUC達0.83和0.93,并且YWHAG能夠預測從認知正常向前驅(qū)期AD的轉(zhuǎn)化,以及從MCI(輕度認知障礙)向AD癡呆的轉(zhuǎn)化,與AD核心病理標志物以及認知能力下降密切相關。
除了診斷,這些蛋白是否可用于AD預警呢?程煒所在的研究團隊還從臨床角度考察了這些蛋白質(zhì)的縱向預測能力,即通過基線時蛋白質(zhì)水平的高低將人群分為兩組,可觀察到高水平組隨著時間推移,發(fā)病人群逐漸增加,從而證明了這些蛋白質(zhì)可作為早期預警指標。另外,研究還探討了這些蛋白質(zhì)與β淀粉樣蛋白、Tau蛋白等AD經(jīng)典病理標志物的關聯(lián),發(fā)現(xiàn)它們高度相關。
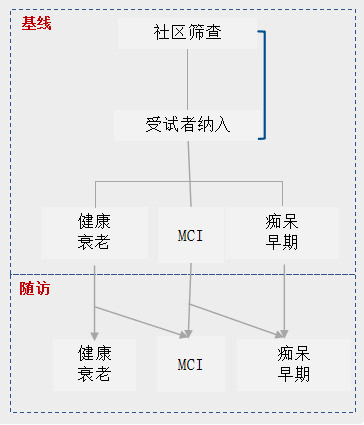
基線與隨訪 圖片來源:演講者供圖
“不過,這些新的生物標志物和此前的生物標志物的性能差不多,更重要的還是要看它們能不能在血液中被檢測出來。”程煒說。
目前,AD早期診斷可以通過腦脊液檢測或PET(正電子發(fā)射斷層成像)測量進行,但前者是有創(chuàng)檢測,后者相當昂貴。因此,程煒的團隊將研究對象從腦脊液蛋白質(zhì)組轉(zhuǎn)到血液蛋白質(zhì)組,希望找到公眾更容易接受的檢測方式。
好消息是,研究團隊基于11種血液蛋白構(gòu)建的AD風險預警模型,AUC達到0.85左右;生存分析表明GFAP、NEFL、GDF15等蛋白與各種癡呆發(fā)病相關,且這些蛋白質(zhì)在AD患者發(fā)病前15年就已表現(xiàn)出顯著差異。而且,GFAP這一蛋白質(zhì)還顯示出特異性,僅對AD有預測價值。
基于此,研究團隊構(gòu)建了基于機器學習的預測模型,通過效果值方法對蛋白質(zhì)的重要性進行排序,結(jié)果與臨床發(fā)現(xiàn)高度一致。最終,團隊構(gòu)建的預測模型能夠提前15年預測AD發(fā)病風險,對于AD的蛋白組合,聯(lián)合模型AUC可以達到0.854,對于癡呆AUC可以達到0.841,為社區(qū)篩查提供了潛在可能。
但程煒指出,蛋白質(zhì)的檢測成本仍然比較昂貴,為了使模型更便于推廣,團隊基于UK Biobank的50萬人縱向隨訪數(shù)據(jù),繪制了AD臨床診斷前15年多維度表型的變化軌跡,并基于顯著變化的指標構(gòu)建了簡易的AD預測模型,該模型僅依賴于血漿GFAP與簡便的人口學特征,AUC就可以達到0.872。
“其實除了AD,我們還希望將模型推廣到其他老年疾病的風險預警。”程煒透露,目前團隊還構(gòu)建了基于血液蛋白組學的多疾病早期預測模型、基于神經(jīng)網(wǎng)絡的蛋白風險預測模型,他認為未來通過數(shù)字化平臺和大模型框架,可以實現(xiàn)更多社區(qū)人群、更多疾病的早期預警和干預。
轉(zhuǎn)載來源:每日經(jīng)濟新聞 作者: 林姿辰

 京公網(wǎng)安備 11010802028547號
京公網(wǎng)安備 11010802028547號